Este estudio fue aprobado por la Junta de Revisión Institucional de nuestra institución y se eximió del requisito de consentimiento informado debido a la naturaleza retrospectiva del estudio.
Fuentes de datos
Para desarrollar un algoritmo de segmentación multiorgánica para tomografías computarizadas abdominales, se utilizó un conjunto de datos que constaba de 95 exploraciones DECT que se asignaron aleatoriamente a uno de los siguientes tres grupos: (1) conjunto de entrenamiento (n = 75); (2) conjunto de validación (n = 10); y (3) el conjunto de pruebas de entrenamiento (n = 10). Además, para evaluar el rendimiento del algoritmo, se recopilaron por separado dos conjuntos de datos externos independientes que constan de 30 pruebas DECT (conjunto de pruebas DECT) y 30 pruebas de CT de energía única (SECT) (conjunto de pruebas SECT). A continuación se proporcionan detalles específicos de los conjuntos de datos utilizados en este estudio.
Conjunto de datos de TC de energía dual
Para desarrollar el algoritmo, recopilamos 95 tomografías computarizadas abdominales dinámicas con contraste de 95 pacientes, realizadas entre abril de 2020 y septiembre de 2020, utilizando dos dispositivos DECT diferentes: SOMATOM Force, Siemens Healthineers (n = 49) e IQon Spectral CT, Philips. Atención de Salud (n = 46). Los criterios de inclusión para el conjunto de datos DECT fueron generalmente pacientes ambulatorios adultos sanos asintomáticos sin lesiones focales, excluyendo pequeños quistes en órganos diana, incluidos el hígado, el bazo, el riñón derecho (RK), el riñón izquierdo (LK) y el páncreas.
Para probar externamente el rendimiento del algoritmo, se realizó un conjunto de pruebas DECT temporalmente independientes de 30 pruebas DECT de enero a febrero de 2021 utilizando dos dispositivos DECT diferentes (SOMATOM Force, Siemens Healthineers [n = 15]y IQon Spectral CT, Philips Healthcare [n = 15]) ha sido recopilado.
Se utilizaron imágenes de fase venosa portal de energía dual (DE-PVP) como conjunto de datos para imágenes de TC poscontraste. Las imágenes DE-PVP cubrieron esta área desde la parte superior de la cúpula hemidiafragmática superior hasta la espina ilíaca anterosuperior o el nivel de la parte superior del muslo. Los parámetros de adquisición y reconstrucción de TC utilizados para cada escáner se muestran en la Tabla complementaria 1. Las imágenes VNC correspondientes se utilizaron como conjunto de datos para las imágenes de TC sin contraste. Estas imágenes VNC se generaron a partir de datos sin procesar DE-PVP utilizando sistemas de posprocesamiento dedicados, como Syngo.via para SOMATOM Force e IntelliSpace Portal para IQon Spectral CT.
Conjunto de datos CT de energía única
Para el conjunto de datos SECT, se recopilaron 30 tomografías computarizadas abdominales con contraste dinámico de 30 pacientes (conjunto de pruebas SECT), que se adquirieron utilizando tres escáneres SECT diferentes en nuestra institución entre enero de 2021 y febrero de 2021: Revolution, GE Healthcare (n = 9 ), Definición de SOMATOM, Siemens Healthineers (n = 11) y TIC, Philips Healthcare (n = 10). Cada tomografía computarizada incluyó imágenes TNC e imágenes PVP de energía única (SE-PVP). Los criterios de inclusión para el conjunto de datos SECT fueron idénticos a los utilizados para el conjunto de datos DECT. El rango de exploración tanto para SE-PVP como para TNC fue desde la parte superior del hemidoma superior hasta la espina ilíaca anterosuperior o el nivel de la parte superior del muslo. Los parámetros de CT detallados para cada escáner se enumeran en la Tabla complementaria 1.
Crea pegatinas 3D para miembros
Se generaron etiquetas de órganos 3D para hígado, bazo, RK, LK y páncreas en conjuntos de datos DECT y SECT. Para mejorar la eficiencia del proceso de etiquetado, inicialmente se utilizó un software de segmentación disponible comercialmente (MEDIP PRO v.2.4.0, MEDICALIP Co. Ltd., Seúl, Corea) para segmentar los órganos primarios. A continuación, un radiólogo abdominal certificado (SKJ, con 9 años de experiencia clínica en la interpretación de tomografías computarizadas abdominales) realizó manualmente la corrección de vóxeles de las etiquetas sin procesar y las colocó como anotaciones reales. Para DECT, las etiquetas de órganos 3D se confirmaron en imágenes DE-PVP. Estas etiquetas confirmadas luego se transfirieron directamente a las imágenes VNC correspondientes según la coincidencia espaciotemporal (Figura 1 complementaria). Por el contrario, para SECT, las etiquetas de órganos se generaron por separado para las imágenes SE-PVP y TNC. Las etiquetas TNC se crearon haciendo referencia a imágenes PVP del mismo paciente para garantizar la precisión en el proceso de segmentación de órganos.
Algoritmo 3D nnU-Net para segmentación de múltiples órganos
Desarrollo de algoritmos
Se desarrolló un algoritmo de segmentación de múltiples órganos para tomografías computarizadas abdominales utilizando la arquitectura 3D nnU-Net (Figura 1), que es un modelo CNN muy avanzado conocido por su gran rendimiento y eficacia en la segmentación de imágenes médicas.13. Para desarrollar el algoritmo, se utilizó un conjunto de datos que comprende 20.020 cortes de imágenes PVP y sus correspondientes imágenes VNC de 85 pruebas DECT con anotaciones de verdad sobre el terreno. En lugar de entrenar exhaustivamente el algoritmo de segmentación, la red está diseñada para tomar las regiones de los órganos internos predichas utilizando el algoritmo de segmentación de la composición corporal.14 como datos de entrada, creando cinco clases correspondientes a regiones segmentadas en hígado, bazo, RK, LK y páncreas (Figura 2 complementaria). El algoritmo de segmentación de la composición corporal que utilizamos para predecir las áreas de los órganos internos funciona proporcionando automáticamente una segmentación volumétrica de los componentes del cuerpo en siete categorías: piel, huesos, músculos, grasa visceral abdominal, grasa subcutánea, sistema nervioso central y órganos internos vascularizados. Investigaciones anteriores han informado que los coeficientes de similitud de dados (DSC) superan 0,94 para este algoritmo.14. Adoptamos esta estrategia de desarrollo, utilizando regiones de órganos internos como entrada, para reducir el tiempo de inferencia evitando estratégicamente regiones de órganos no internos, reduciendo así la carga computacional y reduciendo la incidencia de falsos positivos.
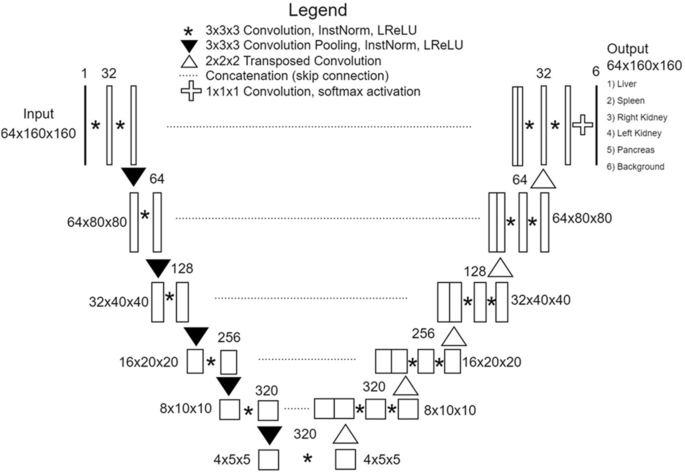
Arquitectura del algoritmo de segmentación de múltiples órganos basado en 3D nnU-Net.
Las estrategias de preprocesamiento y los hiperparámetros de red se adaptan para mejorar el rendimiento de nnU-Net. Como parte del paso de preprocesamiento, se cambió el tamaño de los volúmenes 3D para que coincidan con las distancias objetivo de 0,66796875, 0,66796875 y 2. La configuración final del tamaño del parche fue 160 x 160 x 64 y el tamaño del lote fue 2. Durante el entrenamiento, se utilizaron técnicas de aumento de datos, como rotación, escalado, corrección gamma y duplicación. Se utilizaron funciones de pérdida de dados y pérdida de entropía cruzada para entrenar el algoritmo mediante descenso de gradiente estocástico (impulso de Nesterov = 0,99). El programador de tasa de aprendizaje polinomial se inicializa a 0,01. El algoritmo fue entrenado durante 1000 épocas.
Rendimiento del algoritmo de segmentación.
En primer lugar, se evaluó el rendimiento de segmentación del algoritmo utilizando el conjunto de pruebas de entrenamiento, que forma parte del conjunto de datos de desarrollo que no se utiliza para el entrenamiento. Además, se realizaron pruebas externas utilizando dos conjuntos de datos separados (conjunto de pruebas DECT y conjunto de pruebas SECT). Para evaluar el rendimiento de la segmentación, se usaron imágenes DE-PVP y sus correspondientes imágenes VNC en el grupo DECT y se usaron imágenes SE-PVP y TNC en el grupo SECT (Figura 3 complementaria).
Estimación del tamaño del órgano utilizando el algoritmo desarrollado.
Se probó la aplicabilidad clínica del algoritmo desarrollado evaluando su precisión en la medición de volúmenes de órganos en conjuntos de pruebas externos (conjunto DECT y conjunto SECT). El volumen de cada órgano diana (hígado, bazo, RK, LK y páncreas) se midió en función de los resultados de la segmentación 3D obtenidos del algoritmo.
análisis estadístico
La precisión del algoritmo de segmentación de órganos 3D nnU-net desarrollado se evaluó para cada fase de órgano e imágenes comparando las máscaras derivadas del algoritmo con las máscaras reales del terreno. Coeficiente de similitud de dados (DSC)15 Se utilizó como escala de evaluación. Además, se compararon los valores de DSC para diferentes fases de imagen entre los grupos de prueba DECT y SECT. Específicamente, los valores de DSC entre DE-PVP y VNC en DECT y entre SE-PVP y TNC en SECT se calcularon utilizando R-Examen. Además, se compararon los valores de DSC entre los escáneres de TC que utilizan Mann-Whitney. sh Prueba o prueba de Kruskal-Wallis.
Para el tamaño de los órganos, la correlación entre el tamaño estimado obtenido por el algoritmo y el tamaño real de cada órgano se evaluó mediante el análisis de correlación de Pearson. La concordancia entre los volúmenes estimados y reales se evaluó mediante el coeficiente de correlación intraclase (ICC) y el análisis de Bland-Altman. Los valores de ICC se interpretaron utilizando los siguientes criterios: ≥ 0,90 indica una concordancia excelente; ≥ 0,75 a < 0,90, buena concordancia; ≥ 0,50 a < 0,75, acuerdo moderado; y <0,50, mal acuerdodieciséis. Los límites de acuerdo (LOA) del 95% de Bland-Altman se expresaron como un porcentaje de los valores medidos. El error porcentual del volumen estimado del algoritmo en comparación con el volumen real del terreno se calculó mediante la fórmula [(algorithm-estimated) − (ground truth)]/(verdad fundamental) x 100 (%). Una estimación precisa del tamaño se definió como una evaluación que se desvía no más del 10% del tamaño real del terreno. En consecuencia, la sobreestimación se definió como una desviación de >10% del volumen de verdad fundamental, mientras que la subestimación se definió como una desviación de <-10% del volumen de verdad fundamental.
Todos los análisis estadísticos se realizaron con MedCalc versión 19.4.0 (software MedCalc, Ostende, Bélgica). a s La significación se fijó en <0,05.

«Organizador. Escritor. Nerd malvado del café. Evangelista general de la comida. Fanático de la cerveza de toda la vida.







