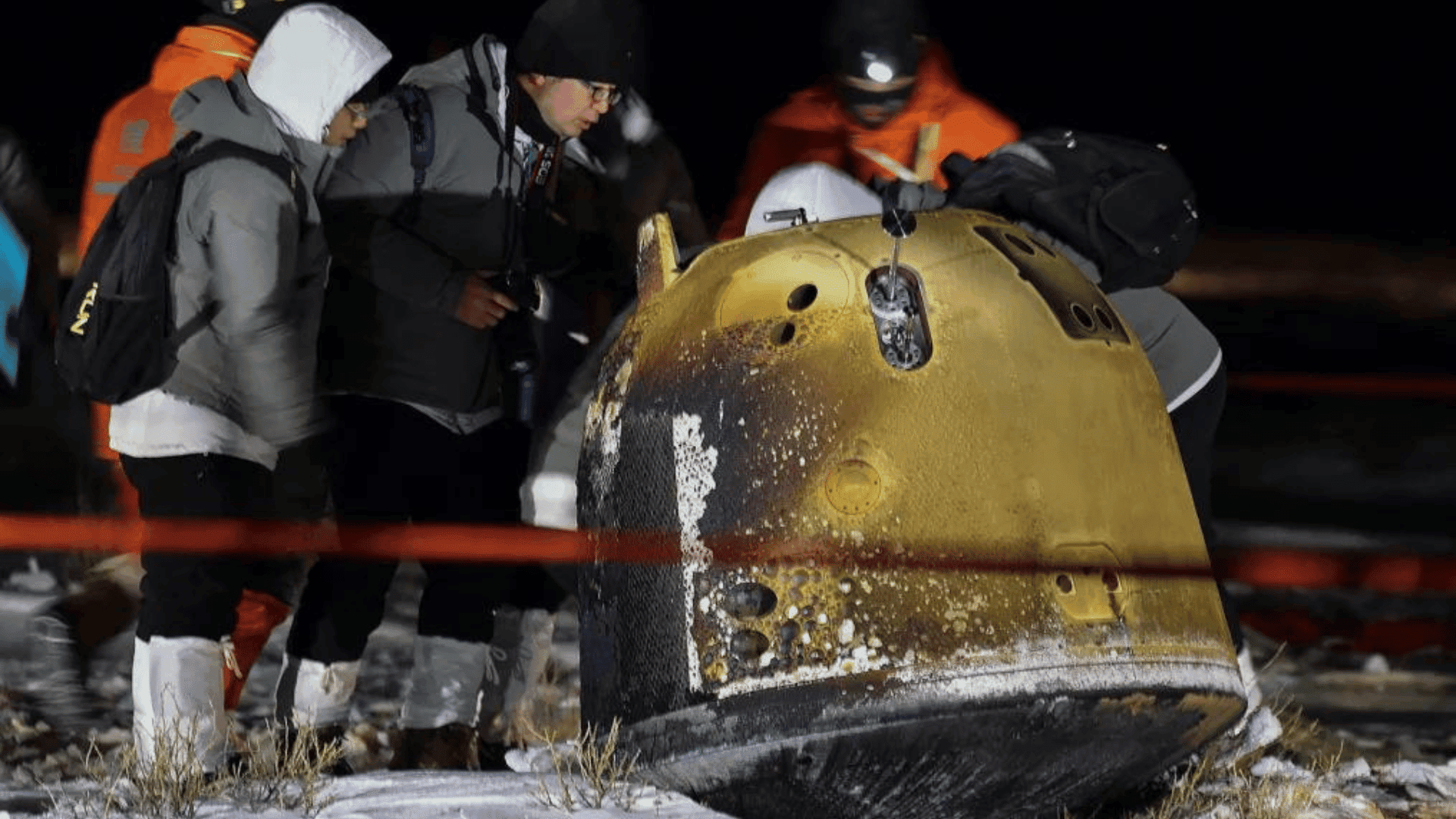En un estudio reciente publicado en procedimientos de la Academia Nacional de Cienciaslos investigadores mostraron la contención de bacterias biológicas y la ingeniería sintética.
Los graves peligros que plantean las enfermedades bacterianas resistentes a los medicamentos y los recientes avances en biología sintética han suscitado un gran interés en los fagos transgénicos con potencial terapéutico. Hasta la fecha, muchos estudios de fagos modificados se han limitado a pruebas de concepto (POC) o investigaciones fundamentales que utilizan fagos con genomas relativamente cortos o «poblaciones de visualización de fagos». Además, no se implementaron precauciones para asegurar una traducción efectiva de la aplicación práctica.
sobre estudiar
El estudio actual creó una plataforma para la ingeniería de fagos recombinantes y libres de células.
Primero, el equipo buscó reiniciar varios fagos de tipo salvaje (WT) que infectaban bacterias Gram-negativas y micobacterias acidorresistentes utilizando los genomas recolectados. en el laboratorio. Se usó la reacción en cadena de la polimerasa (PCR) para amplificar el genoma completo del fago ensamblado obtenido a partir del ácido desoxirribonucleico (ADN) o del genoma molde. Se desarrollaron cebadores para generar regiones homólogas de 28 pb a 65 pb en cada fragmento de PCR flanqueante. Estas piezas se agregaron y electrificaron en huéspedes bacterianos, reactivando los fagos.
El equipo desarrolló fragmentos que permitieron unir los extremos de los fagos reactivados derivados de los genomas circulares. El colifago lambda se reactivó eficazmente a partir de cinco fragmentos en el fago P22 de Salmonella y E. coli, junto con cuatro fragmentos presentes en S. Typhimurium LT2. También se reinició el modelo de micófago D29, que infecta a Mycobacterium spp, que es ácido resistente y tiene un genoma alto en guanina y citosina. Se produjeron cinco segmentos de ADN para crear un genoma circular.
consecuencias
El modelo de colifago T7, que contiene un genoma lineal, mostró secuencias lineales en el momento de la replicación y se utilizó para evaluar la estrategia de estudio. El genoma linealizado se construyó a partir de cuatro fragmentos mediante la reacción en cadena de la polimerasa (PCR) y luego se apiló electroforéticamente en E. coli para reactivar el fago T7. Se predijo que el colifago T3, el fago gh-1 de Pseudomonas y el fago SP6 de Salmonella tenían estructuras genéticas y ciclos de vida similares a los de T7. El equipo también logró regenerar T3 a partir de fragmentos de ADN de E. coli, SP6 a partir de fragmentos de ADN de Salmonella Typhimurium LT2 y gh-1 a partir de fragmentos de ADN de Pseudomonas putida. Estos resultados mostraron que las pautas de diseño y la metodología del estudio fueron efectivas para reiniciar los fagos de los genomas lineales ensamblados.
El mycophage D29 funcional, el mycophage B1 no etiquetado y el GS4E se reiniciaron a partir de M. smegmatis mc2 155. Mycophage TM4 se construyó a partir de seis segmentos de ADN producidos químicamente en función de los datos de secuenciación disponibles públicamente. Los fragmentos de ADN se amplificaron para demostrar una superposición de 28 pb a 38 pb con fragmentos adyacentes, se purificaron y luego se empalmaron. en el laboratorio. El genoma se insertó en M. smegmatis mc2 155. Se reinició el complejo sintético activo TM4, cuyo genoma se produjo bioquímicamente. MiSeq secuenció todo el genoma sintético de TM4 y demostró ser 100 % idéntico al TM4 indicado, lo que indica una reproducción eficiente del fago a partir de los datos de secuenciación.
El Phage T7 premium está bien seleccionado como modelo para la realización de POC. La máquina T7 seleccionó una secuencia de señal de empaquetamiento (Pac) para ‘encapsular’ o ‘almacenar’ su genoma en el autosoma de la cabeza T7. Pac consta de PacB y PacC. La pequeña subunidad gp18 se une al complejo gp19-prohead para trasladar el ADN a la cabeza del fago. Después del empaquetamiento de la cabeza, el equipo observó que gp19 se escinde en PacC en el genoma del fago T7 secuenciado, lo que lleva a la maduración de la cabeza T7.
A continuación, se generó un plásmido que contenía Pac y su ‘gen favorito’ (yfg) y se inyectó en Escherichia coli. La electroporación del genoma en células de Escherichia coli que contienen plásmidos dio como resultado la producción de moléculas transformantes sintéticas dependientes de T7 (T7Pac). En la célula, el genoma Pac-null generó viriones de progenie antes del empaquetamiento del plásmido que contenía Pac en partículas de cabeza, produciendo T7Pac-yfg. Usando este método, el equipo sintetizó T7Pac-lacZ, un plásmido que alberga lacZ. El lisado se combinó con E. coli deficiente en lacZ, seguido de placas con placas LB X-gal. Por lo tanto, 1,6 x 104 Se generaron UFC/ml de T7Pac-lacZ, y todas las colonias formadas eran azules. En el césped bacteriano, T7Pac-lacZ no desarrolló placas, lo que indica contención biológica.
El equipo construyó el genoma del fago SP6 menos el gen que codifica la proteína de la cápside 31. El genoma se insertó en la cepa LT2 de S. Typhimurium, que expresa el gen 31. Mediante PCR, el gen 31 se eliminó del SP6 sintético.Δ cabeza confirmado. Se obtuvo un SP6 de alto calibreΔ cabeza El lisado desarrolló placas en la hierba LT2 que expresan el gen 31, que no estaba presente en los progenitores LT2.
En general, los resultados del estudio mostraron la generación de fagos naturales y manipulados que infectan bacterias Gram negativas y micobacterias acidorresistentes por en el laboratorio Ensamblaje del genoma.

«Erudito en viajes incurable. Pensador. Nerd zombi certificado. Pionero de la televisión extrema. Explorador general. Webaholic».